基因组装神器—MoClo-YTK 模块化酵母克隆工具包
构建酵母多基因表达系统时,你是否面临这些挑战:启动子/终止子活性不稳定,重复实验耗时耗材?多片段组装效率低,Gibson克隆成本居高不下?实验室间元件不兼容,合作研究难以标准化?
酵母基因组装不用“东拼西凑”,一起来看看这个“基因组装神器”—MoClo-YTK模块化酵母克隆工具包!
MoClo-YTK如何成为实验室“基因组装神器”?
MoClo-YTK(Modular Cloning Yeast ToolKit)是一种专为酵母设计的模块化克隆工具包,用于高效、灵活地组装多基因遗传回路或代谢通路。它结合了Golden Gate Assembly的高效拼接方法和标准化模块的设计理念,极大地简化了复杂多基因构建的流程。
该工具包含有一组96 个已验证的高活性启动子、终止子、筛选标记等标准化元件,确保基因表达稳定性和可重复性。其可用于自下而上分层组装单基因和多基因构建体,供酿酒酵母表达;其设计也兼容基于重组的克隆,如酵母体内组装、独立连接克隆(SLIC)或Gibson组装。按标准流程,可在三天内从 PCR 模板构建多基因质粒。MoClo-YTK广泛适用于合成生物学研究、酵母基因组工程、代谢通路优化、多基因表达系统构建等领域。
MoClo-YTK 核心特点?
模块化构建:所有元件遵循BglBrick标准,兼容国际合成生物学通用格式(如MoClo、GoldenBraid),支持跨实验室元件共享。开放架构设计,用户可灵活添加自定义元件或接入第三方工具(如Tom Ellis实验室的MYT升级工具包)。
Golden Gate Assembly 技术:基于Golden Gate技术,单次反应可完成多片段(5-10个)组装,成功率>90%,实验周期缩短50%以上。
酵母优化:模块针对酵母表达系统优化,包含酵母特异的启动子(如 TEF1、GAL1)、终止子、筛选标记(如 URA3、HIS3)等。支持单拷贝或多拷贝整合到酵母基因组,或作为游离型质粒维持。
全流程验证:每个DNA元件均经过启动子强度、终止子效率、筛选标记稳定性等实验验证。
可扩展性强:用户可根据需求自定义和扩展模块,增加新的调控元件、代谢途径等。与植物(MoClo-Plant)、哺乳动物细胞等其他MoClo系统兼容,便于跨物种研究。
低成本兼容性:适配常规实验室设备,普通PCR仪、电泳装置即可完成实验,无需特殊仪器,试剂耗材成本较传统Gibson组装法降低70%。
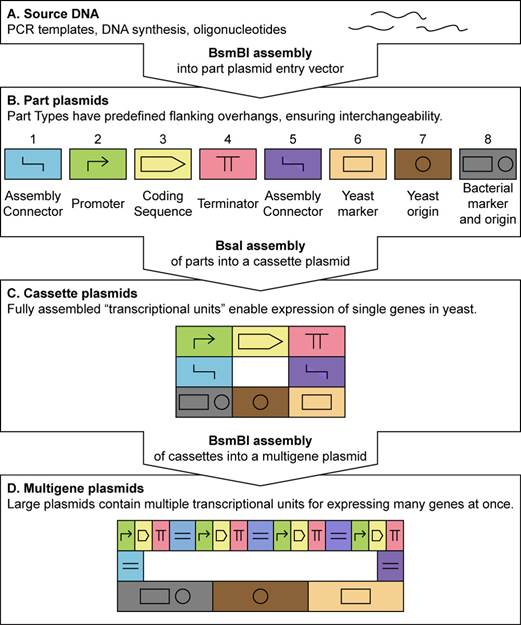
基于 MoClo 的标准化分层装配策略
(图片来源https://pubs.acs.org/doi/10.1021/sb500366v)
MoClo-YTK如何赋能研究?
MoClo-YTK作为先进的酵母模块化克隆工具包,通过"标准化元件库+高效Golden Gate组装"的技术组合,正在革命性地推动前沿研究领域的发展。
合成生物学:构建复杂基因回路
在合成生物学领域,该工具包使复杂基因回路的构建周期缩短70%:以重金属生物传感器为例,研究人员仅用3天就完成了包含响应元件、阻遏蛋白和报告基因的AND逻辑门组装,而传统方法需要2周以上,相关成果已发表于《ACS Synthetic Biology》(2018)。
代谢工程:高效优化产物合成途径
代谢工程应用则展现出更惊人的提升,通过预验证的启动子库精准调控代谢流,β-胡萝卜素生产菌株的产量提升3-5倍,达到工业化生产要求的1.2g/L(《Journal of Industrial Microbiology and Biotechnology》,2020)。
MoClo-YTK是高效的模块化酵母克隆工具包,基于Golden Gate技术实现高成功率多基因组装。包含96个标准化元件,适用于代谢通路优化、酵母工程与合成生物学研究,兼容多种克隆方法,支持快速构建与低成本应用。这些突破性进展与MoClo-YTK两大核心技术优势密不可分:其一,96个已验证标准化元件(涵盖10种强度启动子、8种低泄露终止子)确保实验可重复性;其二,Golden Gate组装技术实现5-10个片段的一步拼接,成功率>90%。该工具包已获得全球近600篇学术文献引用,并被多家生物技术企业用于加速合成生物学、生药物开发进程,充分验证了其在基础研究和产业转化中的双重价值。
MoClo-YTK 模块化酵母克隆工具包详情
|
Cat. NO. |
Plasmid Name |
Part Type |
Part Description |
E.coli Marker |
|---|---|---|---|---|
|
SBDP250001 |
pYTK001 |
entry vector |
Part Plasmid Entry Vector |
Chloramphenicol |
|
SBDP250002 |
pYTK002 |
1 |
ConLS |
Chloramphenicol |
|
SBDP250003 |
pYTK003 |
1 |
ConL1 |
Chloramphenicol |
|
SBDP250004 |
pYTK004 |
1 |
ConL2 |
Chloramphenicol |
|
SBDP250005 |
pYTK005 |
1 |
ConL3 |
Chloramphenicol |
|
SBDP250006 |
pYTK006 |
1 |
ConL4 |
Chloramphenicol |
|
SBDP250007 |
pYTK007 |
1 |
ConL5 |
Chloramphenicol |
|
SBDP250008 |
pYTK008 |
1 |
ConLS' |
Chloramphenicol |
|
SBDP250009 |
pYTK009 |
2 |
pTDH3 |
Chloramphenicol |
|
SBDP250010 |
pYTK010 |
2 |
pCCW12 |
Chloramphenicol |
|
SBDP250011 |
pYTK011 |
2 |
pPGK1 |
Chloramphenicol |
|
SBDP250012 |
pYTK012 |
2 |
pHHF2 |
Chloramphenicol |
|
…… |
…… |
…… |
…… |
…… |
|
更多详情,咨询support@synbio-tech.com |
||||
注:生物资源类业务属于公益共享,我们销售的不是菌种所有权,菌种属于专利所有者,使用者仅可用于做科学研究,不可做其他用途。产品标价均为培养服务费
更多MoClo-YTK工具包详情
请扫码咨询

Reference:
1. Lee ME, DeLoache WC, Cervantes B, Dueber JE. A Highly Characterized Yeast Toolkit for Modular, Multipart Assembly. ACS Synth Biol. 2015 Sep 18;4(9):975-86. doi:10.1021/sb500366v. Epub 2015 May 1. PMID: 25871405.
2. Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 2008;3(11):e3647. doi:10.1371/journal.pone.0003647. Epub 2008 Nov 5. PMID: 18985154
3. Anderson JC, Dueber JE, Leguia M, Wu GC, Goler JA, Arkin AP, Keasling JD. BglBricks: A flexible standard for biological part assembly. J Biol Eng. 2010 Jan 20;4(1):1. doi: 10.1186/1754-1611-4-1. PMID: 20205762; PMCID: PMC2822740.
4. Ostergaard S, Olsson L, Nielsen J. Metabolic engineering of Saccharomyces cerevisiae. Microbiol Mol Biol Rev. 2000 Mar;64(1):34-50. doi: 10.1128/MMBR.64.1.34-50.2000. PMID: 10704473; PMCID: PMC98985.
5. Laughery MF, Wyrick JJ. Simple CRISPR-Cas9 Genome Editing in Saccharomyces cerevisiae. Curr Protoc Mol Biol. 2019 Dec;129(1):e110. doi: 10.1002/cpmb.110. PMID: 31763795; PMCID: PMC6986324.
6. Kim HJ, Jeong H, Lee SJ. Synthetic biology for microbial heavy metal biosensors. Anal Bioanal Chem. 2018 Feb;410(4):1191-1203. doi: 10.1007/s00216-017-0751-6. Epub 2017 Nov 28. PMID: 29184994.
7. Buli Su, Dandan Song, Fan Yang, Honghui Zhu, Engineering a growth-phase-dependent biosynthetic pathway for carotenoid production in Saccharomyces cerevisiae, Journal of Industrial Microbiology and Biotechnology, Volume 47, Issue 4-5, 1 May 2020, Pages 383–393, doi.org/10.1007/s10295-020-02271-x
8. Ryan OW, Poddar S, Cate JH. CRISPR-Cas9 Genome Engineering in Saccharomyces cerevisiae Cells. Cold Spring Harb Protoc. 2016 Jun 1;2016(6). doi: 10.1101/pdb.prot086827. PMID: 27250940.
 Syno®C 引物合成
Syno®C 引物合成 RNA合成
RNA合成 mRNA合成
mRNA合成 Syno®GS 基因合成
Syno®GS 基因合成 载体构建
载体构建 高通量及DNA文库构建
高通量及DNA文库构建 CRISPR基因编辑平台
CRISPR基因编辑平台 病毒包装
病毒包装 基因测序及分析
基因测序及分析 重组蛋白表达平台
重组蛋白表达平台 抗体工程平台
抗体工程平台 多肽服务
多肽服务 生物信息学分析与设计
生物信息学分析与设计 CRISPR文库
CRISPR文库 合成生物学产品
合成生物学产品 ProXpress蛋白快速检测
ProXpress蛋白快速检测 CRISPR 质粒
CRISPR 质粒





















